基于电子密度驱动的3D分子生成:望石智慧以技术驱动药物研发底层变革
1.1引言
近年来,人工智能驱动药物设计(AIDD)已成为加速新药发现进程的核心技术。然而,传统AIDD方法仍存在一定的局限性。首先,在分子表征方面,主流方法大多依赖于简化分子线性输入系统(SMILES)或二维分子图等符号化表示,然而这些表征方法却缺乏分子在三维空间中的关键结构信息,无法有效模拟其与靶点口袋之间复杂的空间的相互作用。其次,蛋白质数据库中已解析的原子坐标是人为拟合实验电子密度图后得出的静态坐标数据,缺乏反应小分子与蛋白间的动态相互作用及溶剂环境的原始物理信息,使得模型无法学习到关键相互作用信息。在模型架构层面,无论是自回归的方法、图神经网络还是新兴的扩散模型,都常常在分子结构的化学合理性、类药性、可合成性等方面难以取得平衡,易于生成难以合成或结合模式不理想的分子。
正是在这样的背景下,望石智慧的研究工作展现出一条清晰且扎实的技术路线,没有选择在传统方法内逐渐优化,而是开创性地将实验电子密度(Experimental Electron Density, ED)这一未被充分挖掘的物理信息作为其AI模型训练及推理的基石,并结合创新的分子表征方法与模块化的设计系统性地构建了一个从数据表征到生成与评估的完整技术闭环。其研究工作并非孤立的技术点突破,而是一个层层深入、相互支撑的协同体系,旨在从根本上提升AIDD方法生成分子的三维结构合理性、类药性及可合成性。
1.2电子云密度数据应用:首个实验性非共价相互作用数据库上线
实验电子密度在识别化合物与蛋白间弱相互作用和捕捉动态相互作用方面具有显著特点,通过分析不同分辨率的电子密度图,可以有效增强非关键相互作用信号的信噪比,有助于挖掘二者潜在的相互作用。望石团队首次实现了将实验电子密度直接应用于蛋白质-配体复合物的非共价相互作用分析。具体而言,他们首次系统性地利用蛋白质晶体数据库(Protein Data Bank, PDB)中的实验电子密度数据,构建了全球首个实验性非共价相互作用数据库(ExptNCI Database),该研究基于电子密度梯度理论,从蛋白质数据库中超过12,000个复合物的实验电子密度图中提取了约20万个相互作用的电子密度鞍点,这些鞍点被视为非共价相互作用的直接实验证据,并利用电子密度的时间平均特性成功捕捉到了由侧链旋转等运动导致的动态相互作用模式(图1)。
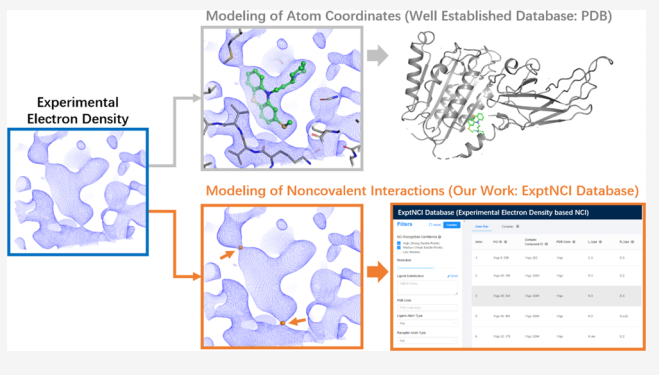
图 1. ExptNCI数据库展示
1.3从数据到模型:以实验性数据指导分子生成
1.3.1模型架构设计
有了相应的电子密度数据之后,望石团队开始利用实验电子密度作为训练数据,设计基于靶蛋白口袋结构生成类药三维分子的方法。其工作原理如图2所示,在初始阶段,利用生成对抗网络(GAN)以口袋电子密度数据为输入以学习口袋-配体互补性并生成配体的电子密度数据。随后,由矢量化变分自编码器(VQ-VAE)来捕获分子拓扑与电子分布特征;再通过PixelCNN以生成的配体的电子密度数据为条件在隐空间中采样,从而生成多样性较高的配体的电子密度数据(图2)。最后,将这些电子密度数据还原成分子,并通过可合成性与类药性打分筛选掉不满足条件的分子,确保化学结构的合理性。
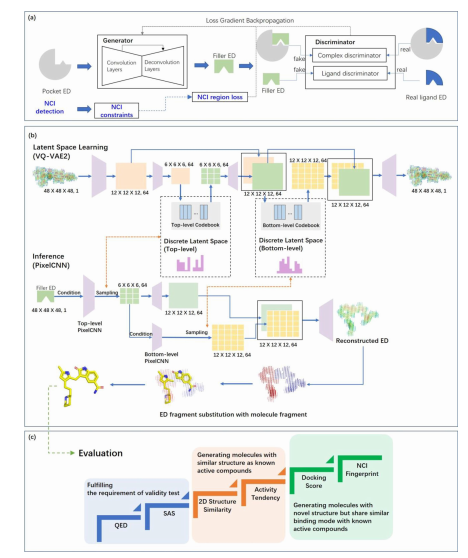
图 2. 基于电子密度的分子生成模型设计架构
1.3.2实验验证与结果
望石团队将该方法针对于三个重要药物靶点(HPK1(造血祖细胞激酶1)、3CLpro(SARS-CoV-2 主蛋白酶)、VDR(维生素D受体))进行了分子生成,并基于包含8000余个文献报道活性化合物的参考数据集进行了全面评估。评估维度涵盖化学有效性、化学空间分布的多样性,以及与参考活性化合物在分子结构及口袋结合模式方面的相似性等方面。
研究结果表明,该方法不仅能够生成与已知活性化合物结构相似的分子,还可生成具有新颖骨架且保持关键结合特征的化合物,显示出其在构建有效虚拟筛选库方面的潜力。此外,该方法所生成的配体电子密度信息,同样也为基于片段的药物设计提供了线索。
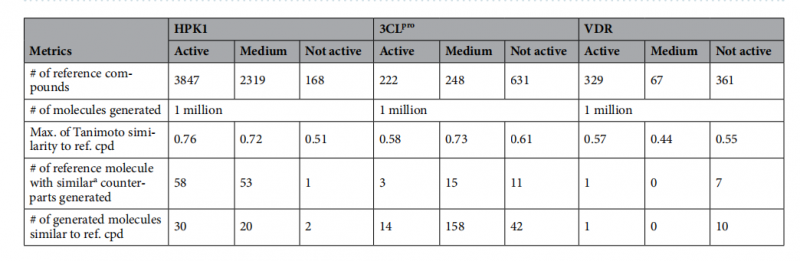
图 3 生成分子与参考活性分子的相似性比较
1.4电子密度在虚拟筛选中的应用:独创ExptGMS方法
1.4.1 ExptGMS设计思路
为了解决传统虚拟筛选方法中因依赖静态、不完整的蛋白质结构模型而导致的打分函数准确率受限的问题,望石团队以电子密度图为基础,仅保留等值面水平高于零的电子密度区域,以此排除实验噪声。此外,还在蛋白质结合口袋内部及周边空间构建出可用于定量评估的三维评分网格,并根据配体原子是否占据电子密度网格来设计相应的打分函数(图4)。

图 4. 实验电子密度图提供的潜在信息
1.4.2 ExptGMS方法在实际药物发现中的应用:Covid-19 3CLpro抑制剂的筛选
为验证ExptGMS方法在实际药物发现中的应用价值,该团队针对新冠病毒3CL蛋白酶进行了虚拟筛选。基于该蛋白的晶体结构,首先通过分子对接软件对大规模商业化合物库进行初步筛选,并设定关键相互作用作为对接约束条件。随后,采用实验电子密度图对对接结果利用ExptGMS重新打分,通过联合考虑对接分数和电子密度匹配度的策略,筛选出若干候选化合物进行实验验证,即采用荧光共振能量转移法测定所有候选化合物的酶抑制活性。与此同时,还设置了仅基于对接评分的对照组进行比较。结果表明,采用ExptGMS辅助筛选的策略显著提升了活性化合物的发现效率,实验组中多个化合物表现出显著抑制活性,并且就其筛选结果来看,ExptGMS的结果表现明显优于对照组的结果。这也证明了ExptGMS在真实药物筛选场景中能够有效提升虚拟筛选的成功率和实用性,弥补传统打分函数的不足(图5)。
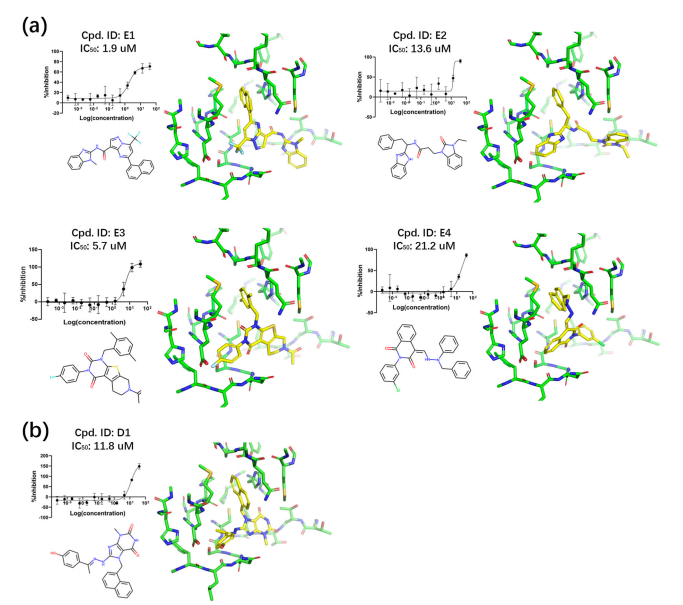
图 5. 生成的Covid-19 3Clpro活性分子展示
1.5语言模型驱动的3D分子生成:兼顾分子拓扑与空间坐标的Lingo3DMol
1.5.1 模型设计
传统的基于序列或者基于分子图的分子生成方法因缺失了分子的三维空间几何信息,往往在生成过程易于产生极不稳定、难以合成的结构。为此,望石团队设计了一款自回归的分子生成方法Lingo3DMol,并开发了基于片段的简化分子线性输入规范(fragment-based simplified molecular-input line-entry system, FSMILES)的新分子表示方法,在编码时不仅引入了原子类型,还同时加入了原子的局部坐标和全局坐标信息。
此外,传统方法在识别与靶点口袋的关键相互作用时存在一定局限性。为此,还训练了一个独立的非共价相互作用(Non-covalent interactions,NCI)预测模型,即预测哪些口袋原子最可能与配体形成哪种类型的相互作用。这样做可以引导它生成更可能通过特定相互作用与靶点紧密结合的分子,极大地提高了生成分子的潜在活性。
为了消解自回归生成过程中院子坐标定位不准确的问题,还采用了两个协同工作的解码器。二维拓扑解码器负责生成FSMILES序列,决定分子的二维连接方式和局部坐标。三维全局坐标解码器负责预测原子在口袋空间中的绝对坐标,从而让模型在生成的过程中让模型主动思考分子的化学结构及其在靶点口袋中的三维姿态,从而生成大量类药性高、易于合成的候选分子,显著提升了基于AI的从头药物设计的实用性和成功率。
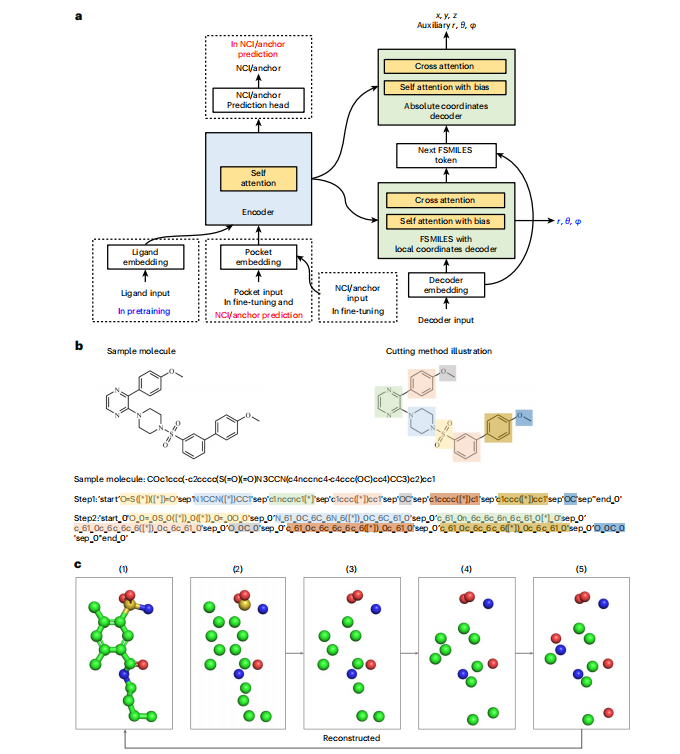
图 6. Lingo3DMol模型设计展示
1.5.2 实验验证与结果
为了验证Lingo3DMol是否可以生成高质量的分子,研究团队从分子指纹相似性与分子对接分数两个维度对生成分子进行筛选分析,结果表明模型可以成功复现与已知活性化合物结构及结合模式高度相似的分子,凸显出其解决实际药物发现难题的能力。
此外,传统分子对接方法受限于其构象采样算法的局限性,常因无法充分探索配体的构象空间而遗漏潜在活性分子。然而该模型通过直接生成与靶点口袋三维空间特征相匹配的配体构象,能够有效识别并恢复这类因构象采样不足而被传统虚拟筛选方法错误排除的活性化合物。可见其在生成活性化合物的成功率、减少假阴性方面具有独到优势,为其在基于结构的药物设计中的应用提供了帮助(图7)。
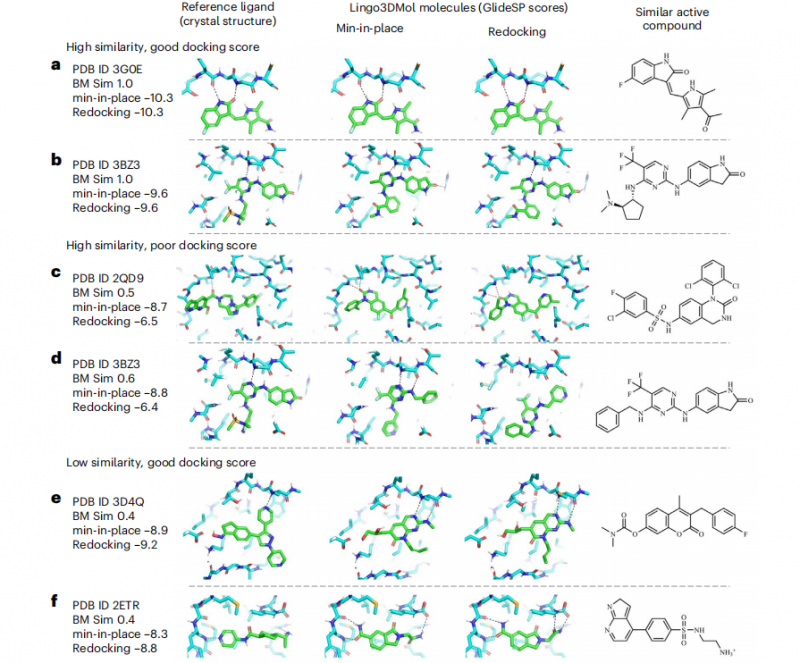
图 7. 生成分子与参考分子的相似性与结合模式分析
1.6总结
传统药物设计方法长期依赖蛋白质结构数据库中的静态坐标信息,然而这些信息无法捕捉蛋白质-配体复合物的动态构象变化,忽略了关键的结合过程信息。若要减少现有药物设计过程中的假阳性率,引入此类如蛋白质-配体复合物的动态构象信息较为重要。
望石智慧的研究团队开创性地将实验电子密度这一物理化学信息引入药物设计全流程,首次设计了新型筛选方法ExptGMS,他们通过将小分子构象与实验电子密度网格进行匹配,因为电子密度图可以反映蛋白质-配体界面上的真实电子分布,可以让模型学习到传统刚性对接所忽略的物理信息,不仅让模型更具可解释性和物理合理性,还有效提升了活性化合物的识别准确率。
此外,还构建了ExptNCI数据库,首次系统地从实验电子密度中提取非共价相互作用信息。由于依赖于实验电子密度图中获取的相互作用的物理信号而非预设规则,因此可以捕获那些传统规则容易忽略的潜在相互作用。
在分子生成方面,团队开发了基于语言模型的三维分子生成方法Lingo3DMol。通过引入片段化SMILES表示与局部-全局坐标编码策略,使得在保持合理三维构象的同时,显著提升了生成分子的类药性与可合成性。然而该方法存在一定的局限性,即生成分子后需要通过分子对接和动力学模拟等计算方法来进一步优化其结合姿势和能量。望石团队以实验电子密度为核心所构建的技术体系代表了现代药物设计方法的一次迈进。通过深度整合实验物理信息与前沿人工智能算法构建了覆盖从靶点分析到分子生成全流程的创新平台,为小分子药物研发提供了创新性的技术。这种数据导向的方法也正成为推动人工智能辅助药物设计领域的新动力,在全球药物研发创新中展现出重要的价值与潜力。
1.7参考文献
Ding K, Yin S, Li Z, et al. Observing noncovalent interactions in experimental electron density for macromolecular systems: a novel perspective for protein–ligand interaction research[J]. Journal of Chemical Information and Modeling, 2022, 62(7): 1734-1743.
Wang L, Bai R, Shi X, et al. A pocket-based 3D molecule generative model fueled by experimental electron density[J]. Scientific reports, 2022, 12(1): 15100.
Ma W, Zhang W, Le Y, et al. Using macromolecular electron densities to improve the enrichment of active compounds in virtual screening[J]. Communications Chemistry, 2023, 6(1): 173.
Feng W, Wang L, Lin Z, et al. Generation of 3D molecules in pockets via a language model[J]. Nature Machine Intelligence, 2024, 6(1): 62-73.